大規模な哺乳類ゲノム書き換え技術
ゲノム合成は、マイコプラズマ、大腸菌などの原核生物や、出芽酵母などの真核生物で実行されています。しかし、哺乳類のゲノム合成は、ゲノムのサイズと複雑さゆえに、まだ実現されていません。
例えば、マウスのゲノムをヒト化することを考えた場合、ヒトにはあってマウスにない遺伝子も存在し、遺伝子発現を制御するノンコーディング領域、更に遺伝子発現などに微妙な影響を与える塩基レベルの違いもあります。また、ゾウのゲノムをマンモスのゲノムに書き換えるといった場合にも、このような技術が必要になります。染色体のDNAを小さく張り換える、塩基レベルで変換するという小技ではなく、ある大きな領域をそっくり置き換えるような技術の開発は重要です。
大きなDNA (100kb以上) アセンブリの技術とCreなどの部位特異的リコンビナーゼの使用の組み合わせは、哺乳動物ゲノムの大規模な操作に有効な方法です。しかし、このような大きなDNAフラグメントのこれまでの方法では、つなぎ目などに痕跡のような配列が残ることが問題です。また、巨大なDNAをクローニングするYAC、BACベクターなどを使って、大きなDNAを精製後、単に組み込んだ場合、その挿入部位が予測できず、位置効果の影響を受けるため、本来の遺伝子の発現パターンが再現されないこともしばしばです。
つまり、つなぎ目の痕跡も残さず、染色体上の狙った場所を広範囲にわたって操作するようなゲノム工学技術の開発が必要になっています。
例えば、その一つの方法が、Big-INというものです。
Brosh R, et al. (2021) A versatile platform for locus-scale genome rewriting and verification. Proc Natl Acad Sci U S A. 118:e2023952118. doi: 10.1073/pnas.2023952118.
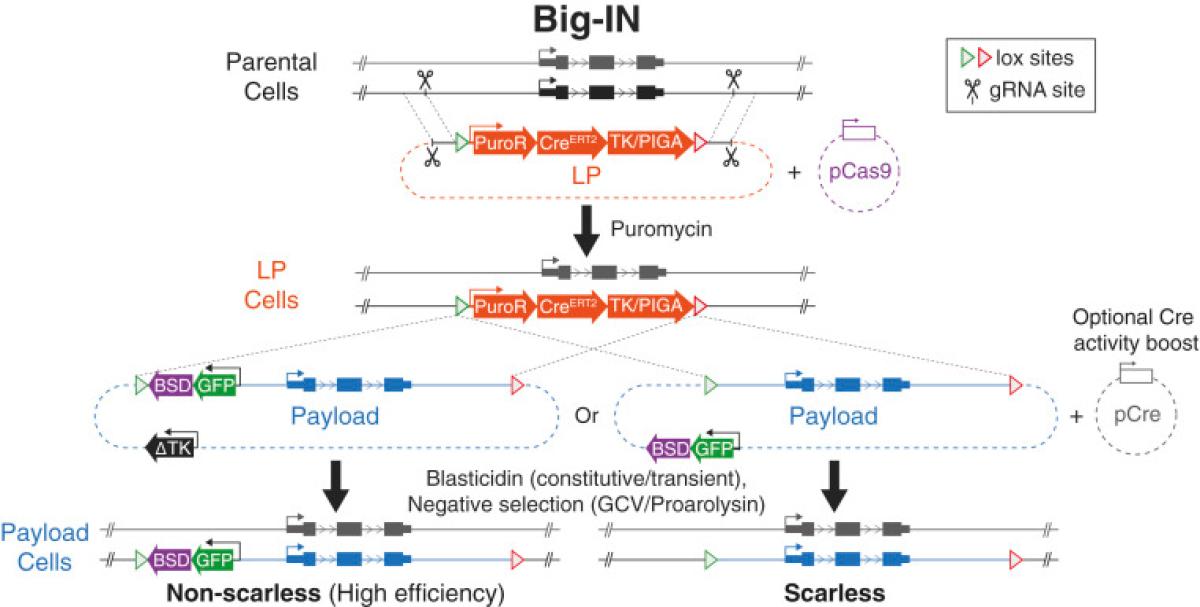
基本的にはES細胞でこれを行うことを意図しています。一見、複雑そうに見えるのですが、いくつかの基本概念がわかっていれば、理解するのは簡単です。CRISPR/Cas9を使ったホモロジー依存組換え(HDR)を使用して、目的遺伝子をLanding pad(LP)に置き換えます。Puromycinは、LPを保有する細胞を選択します。 次に、2対のloxM部位とloxP部位のCre組換えにより、LPとペイロードカセット交換を行い、それをポジティブ、ネガティブ選択していくわけです。
11月1日のNature誌にマウスES細胞のゲノム書き換えを連続的に行う「mSwAP-In」という方法を使って、マウスのp53の遺伝子座Trp53とヒトのSARS-CoV2のレセプターであるACE2を含む遺伝子座をヒト型に書き換えることに成功したという研究が報告されています。
Zhang, W., Golynker, I., Brosh, R. et al. Mouse genome rewriting and tailoring of three important disease loci. Nature (2023). https://doi.org/10.1038/s41586-023-06675-4
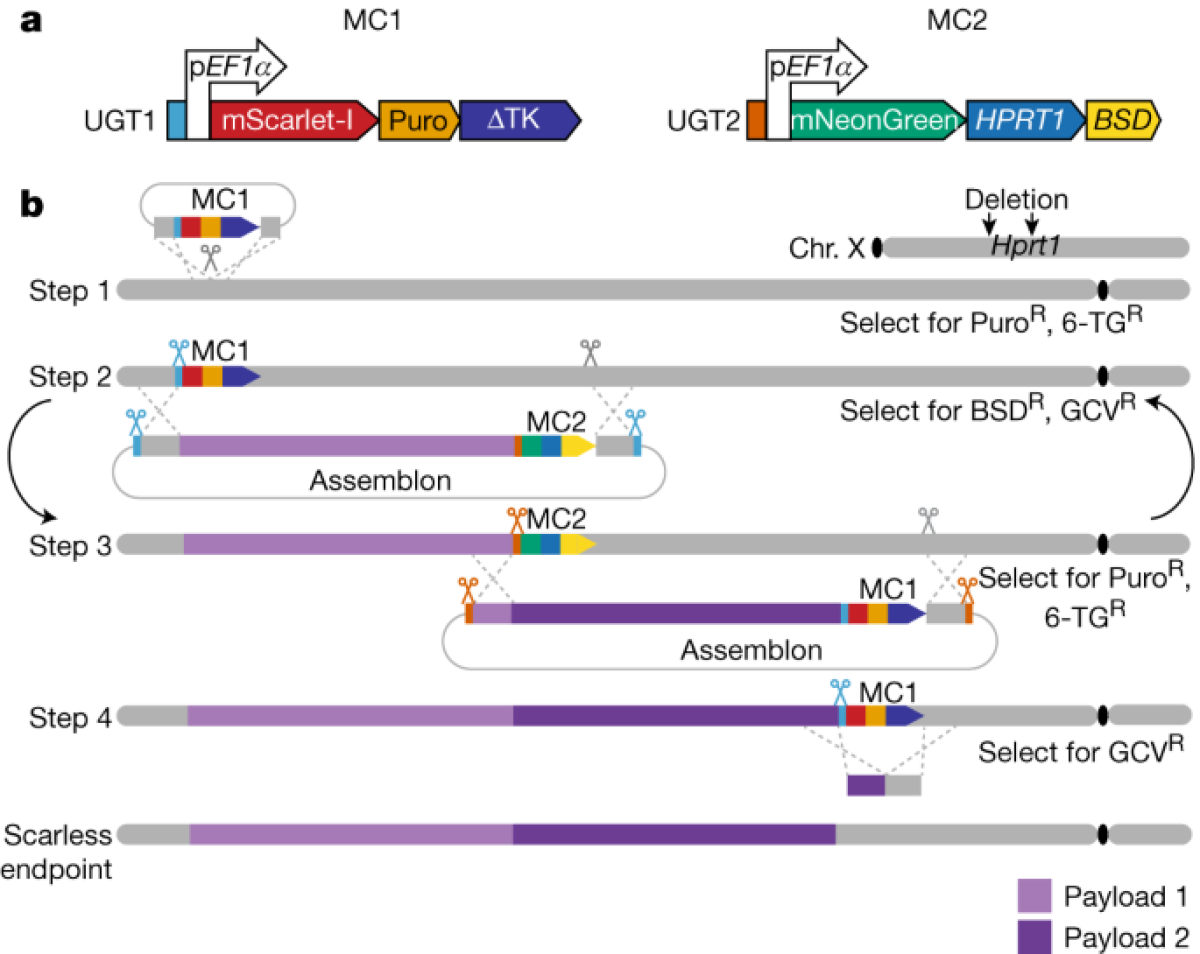
マーカー カセット (MC1 および MC2) は、mSwAP-Inのポジティブ選択およびネガティブ選択の基礎となります。この方法の適用のためには、まず最初に内在性Hprt1を削除する必要があります。標的細胞は、MC1のPuromycin耐性遺伝子を使用したポジティブ選択とMC2のHPRT1に対するネガティブ選択、MC2 (BSD、Blasticidin耐性) のポジティブ選択マーカーとMC1 (Thymidine Kinase切断型) のネガティブ選択マーカーを使用して、順次選択されていきます。
ステップ 1: 目標となる遺伝子座の上流にMC1カセットをCRISPR/Cas9を使ったHDRにより組み込みます 。 灰色のバーはもとの染色体領域です(マウスの配列)。 紫色のバーは合成された入ってくる DNAです(ヒトの配列)。
ステップ 2: HDRのためのMC2 およびペイロードDNAの組み込み。
ステップ 3: ステップ 2 と同じ戦略に従って次のペイロード DNAを組み込み、MC1に戻します。ステップ 2 と 3 は、MC1とMC2を交互に選択することで、無限に繰り返すことができます。
ステップ 4: 最後のMC1またはMC2 の削除(CRISPR-Cas9によるHDR または PiggyBAC を利用して、組換えの痕跡のない細胞を作ることができます)
この方法を使って、116kbおよび180kb のヒトACE2 遺伝子座を使用したマウスを作製したところ、コロナウイルス感染症をより正確にモデル化するゲノムヒト化ACE2マウスができたと報告しています。
前段階として内在性Hprt1を削除したり、薬剤による選択を行う必要があるためES細胞を使う必要があるなど、実際に利用するとなると、かなり面倒そうです。繰り返しているうちに、ES細胞がおかしくなりそうです。個人的な経験では、CRISPR-Cas9などを使ったHDRの効率はかなり高いので、ES細胞の利用をしなくても可能な気がするのですが、CRISPR-Cas9は奇妙なことが起こることもあって、なかなか難しいのかもしれません。
【Twitter】 https://twitter.com/yamagatm3
更新の通知を受け取りましょう

























投稿したコメント